x<-27 # asignamos un valor a x
x>3 # consultamos si x es mayor a 3, el resultado es TRUE[1] TRUELos orígenes del lenguaje R se remontan a los años 90 del siglo XX. La intención de sus creadores, Robert Gentleman y Ross Ihaka del Departamento de Estadística de la Universidad de Auckland en Nueva Zelanda, era elaborar un lenguaje apropiado para el tratamiento estadístico de datos que fuera de acceso libre y de desarrollo colaborativo. Aunque su concepción inicial fue la de ser un lenguaje enfocado al análisis estadístico de datos, hoy día, R constituye un entorno de programación tremendamente versátil, con amplio desarrollo en ámbitos como puede ser la bioinformática, la biomedicina o el análisis de mercados. La entrada dedicada a R en Wikipedia permite hacer un buen repaso sobre la evolución y las características de este entorno. La página oficial del proyecto es conocida como CRAN, acrónimo de Comprehensive R Archive Network, que podríamos traducir como red integral de archivos de R. Se trata de un espacio virtual que permite descargar la versión actualizada de este software, así como los paquetes registrados en él. Un paquete es un conjunto de funciones que, una vez instalado, amplían el lenguaje base dotándolo de nuevas funcionalidades.
La principal crítica a R es que su curva de aprendizaje, -esto es, el perfil de los conocimientos adquiridos frente al tiempo invertido en su estudio-, parece ir lenta al principio. Pero sin duda, el tiempo invertido en aprender este lenguaje en auge suele verse compensado por las prestaciones que ofrece. Como aliciente, destacar el hecho de que R es libre y gratuito, y no por ello menos bueno que los paquetes de análisis estadístico comerciales (cuyo precio suele resultar bastante poco asequible a nivel particular). De hecho, R se situa en los primeros puestos (y en auge) del índice TIOBE, una métrica que mide la popularidad de los lenguajes de programación. Grandes compañías, que bien pueden permitirse pagar la licencia de otros paquetes comerciales, usan R. Este lenguaje no queda al margen del auge de la inteligencia artificial y del análisis masivo de datos. Por otra parte, R interacciona muy bien con aplicaciones comerciales como pueden ser SPSS, Stata, SAS, u hojas de cálculo, lo que permite ampliar su funcionalidad en cuanto al manejo y análisis de datos.
Actualmente, la comunidad de usuarios de R está en crecimiento continuo. Usuarios que generan y comparten recursos analíticos para R y también recursos formativos. Una sencilla búsqueda en la web proporciona rápidamente enlaces a tutoriales, blogs y foros de discusión, como pueden ser R bloggers, Stackexchange, Stackoverflow o datacamp.
Para instalar R, basta con abrir el portal CRAN y seleccionar, en el recuadro inicial de esta página, la versión adecuada para nuestro sistema operativo. Una vez descargado y ejecutado el programa de instalación, ya estará disponible el sistema base R, es decir, el entorno que proporciona la funcionalidad nativa de la aplicación y que podrá ser ampliado mediante la instalación de paquetes. Se alude al lenguaje base para referirse a las funciones implementadas en este entorno sin ampliaciones. Si la instalación se ha realizado con éxito, aparecerá el icono que permite acceder a la interfaz de usuario (GUI, del inglés graphical user interface).
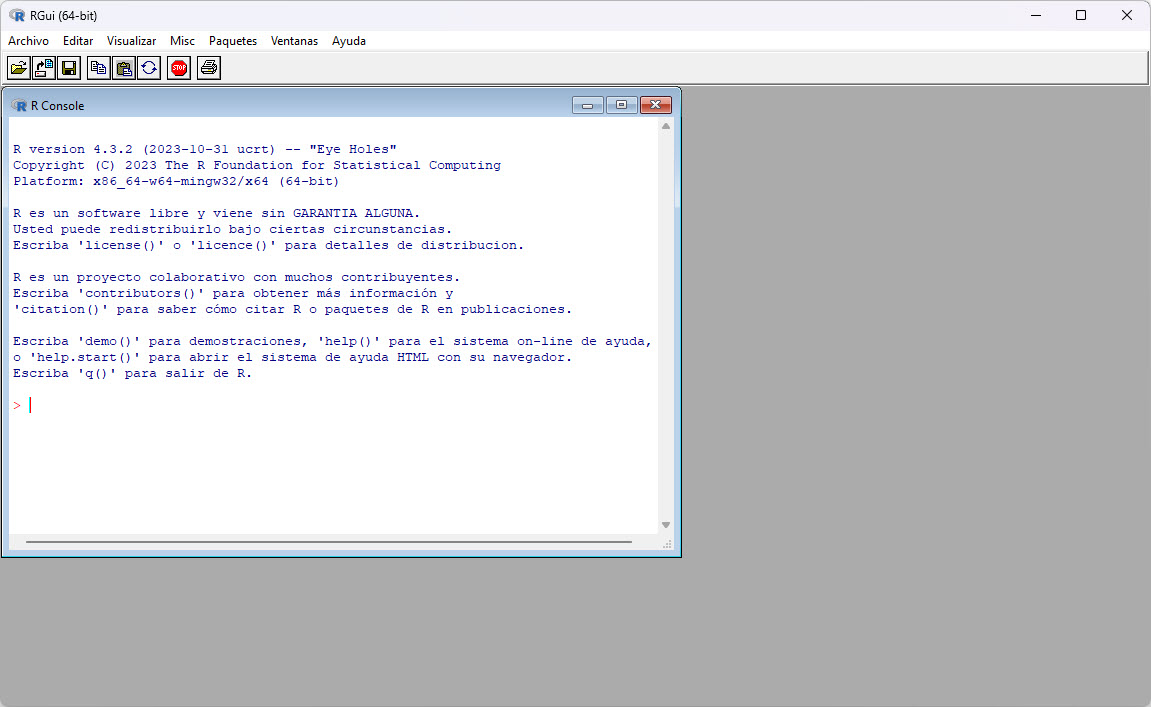
La interfaz, que es muy básica, presenta una ventana con la consola de R y una barra de menús y otra de botones que facilitan la ejecución de algunos procedimientos, como el manejo de archivos o la instalación de paquetes. La consola muestra un mensaje inicial en el que, entre otras cosas, informa de la versión de R que se ha instalado. Tras este mensaje aparece un prompt, es decir, un símbolo que indica que el sistema está preparado para que ejecutar la sentencia cuando la escribamos a continuación de él y pulsemos la tecla intro, como ocurre en el siguiente ejemplo
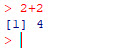
Cuando se ha ejecutado la orden, el sistema muestra el resultado correspondiente (que puede consistir en una o en varias líneas, cada una precedida con la numeración correspondiente entre corchetes) y vuelve a presentar el prompt indicado que está preparado para que se escriba una nueva sentencia.
Podemos definir variables indicando un nombre para la misma y asignándole un valor, o el resultado de una operación, mediante el direccionador <- (se deben escribir los símbolos menor que y el guión sin separación mediante ningún espacio). Estas variables se pueden usar en cálculos subsiguientes. La Figura 3.3 ilustra su uso.

x que almacena el valor 2. Al pulsar la tecla “intro”, R no notifica nada por que el valor 2 se ha enviado a una variable, y no a la salida de resultados. En la segunda línea, se crea una nueva variable y haciendo una operación aritmética con el contenido previamente almacenado en x. Para conocer el valor de y basta con escribir su nombre y pulsar “intro”. Ahora sí que se genera una salida (etiquetada con [1]), ya que no se ha utilizado el direccionador <-.Todas las variables definidas durante una sesión de trabajo son reconocibles en las sentencias siguientes a su definición. Al cerrar el GUI, se pregunta al usuario si desea guardar la sesión. En caso afirmativo, todo lo definido durante la misma estará accesible al volver a abrir la aplicación.
Pero lo cierto es que esta forma de trabajar con R resulta bastante tediosa. Aunque hay paquetes como Rcommander que generan una interfaz más amigable, al estilo de la que tienen la mayoría de los programas estadísticos comerciales, la verdadera potencia de R como entorno de análisis de datos y de programación, se desarrolla con interfaces más elaboradas como Tinn-R o RStudio
El entorno más difundido para interaccionar con R es RStudio.
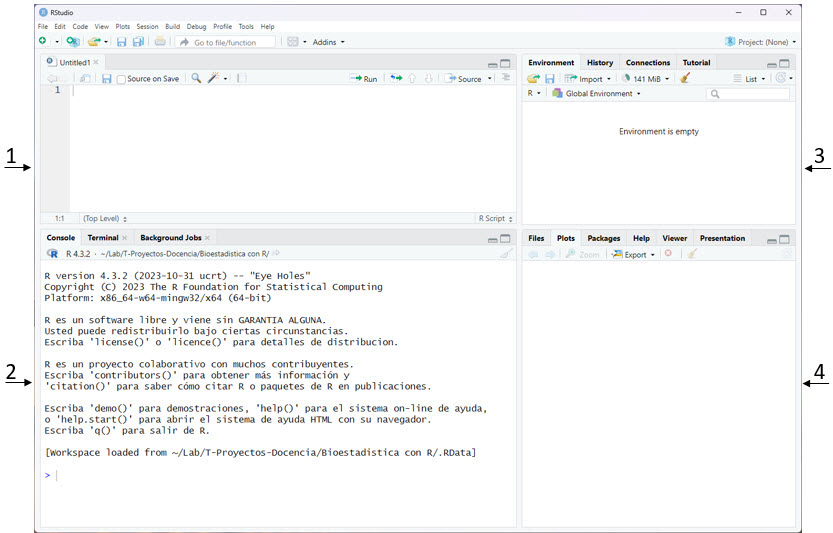
El panel (1) es el editor de archivos de RStudio. Tal y como ilustra el menú “FileFile”, este editor admite un abanico amplio de formatos; R Script, html, C++, Python, Markdown, Quarto, etc. El que nos interesa, de momento, es el formato R Script. Se trata de archivos de texto plano (son visibles y editables con cualquier aplicación, como el bloc de notas de Windows). Un script es un conjunto de sentencias en un determinado lenguaje de programación, de manera que un script de R es un archivo con sentencias y funciones en lenguaje R. Este editor no es simplemente un editor de texto “plano”. Tiene funcionalidades que facilitan la escritura, por ejemplo, al abrir un paréntesis o una llave, se genera automáticamente su cierre, colorea palabras clave del lenguaje (esto es tremendamente útil en programación), detecta y destaca código erróneo, etc. Cada uno de los formatos manejables por este editor genera archivos que tendrán la extensión identificativa apropiada. Los scripts de R tienen extensión “.R”. Para ejecutar la línea de código en la que esté el cursor, o un área de texto previamente seleccionada, es necesario pulsar las teclas data.frame (más adelante se hablará de esta estructura de datos).
El panel (2) es la consola de R. El mismo espacio que muestra la interfaz nativa de R, la R-GUI, que aparece en la Figura 3.1. Igual que en la R-GUI, aparece un prompt (>) indicando que podemos escribir una orden propia del lenguaje y ejecutarla sin más que pulsar la tecla
El panel (3) presenta varias pestañas con funcionalidades diversas. La pestaña “Environment” permite tener un listado de las variables definidas en memoria (las que son accesibles aludiendo a su nombre desde el código). Las tablas de datos, los data.frame, aparecen también en este listado. Si se hace doble click sobre el nombre de un data.frame, este se mostrará en la ventana de edición (1), permitiendo modificar los datos. Además de la pestaña descrita, aparecen History”, para mostrar el historial de sentencias ejecutadas, y “Tutorial” para acceder a un tutorial sobre R. Las otras dos pestañas “Connection” y “Build” son más avanzadas y no se van a abordar de momento.
Antes de ver la forma de manipular datos desde R conviene tener presente cómo se conciben desde el punto de vista informático y su relación con el estadístico. En esta sección, se verá en primer lugar de qué forma se clasifican los datos, para revisar después cómo se estructuran.
En la Sección 2.1 se abordaron los diversos tipos de variables desde el punto de vista estadístico. Como se indicó allí, el tipo de una variable condiciona el tratamiento estadístico que se le puede hacer. A nivel informático también se distinguen diferentes tipos de variable, solo que ahora el tipo, o la clase (en R se usa más este segundo término) se refiere a cómo se almacena la variable y qué operaciones son admisibles con ella. En principio, distinguimos las tres clases siguientes
character)numeric)logical)Una variable de tipo character puede contener cualquier texto, que va a aparecer siempre entrecomillado. Por ejemplo, la asignación x<-"56" almacena en x el texto "56", pero no el valor numérico 56. Si escribimos x+1 lo que obtendremos no será la suma solicitada, sino un mensaje de error. Esto es diferente a escribir x<-56. Ahora x sí que es de tipo numéric y la sentencia x+1 devolverá 57. Las operaciones posibles con las variables alfanuméricas son, entre otras, su ordenación alfabética, la concatenación o su fragmentación en partes de texto, pero no se pueden hacer operaciones aritméticas con ellas. Respecto a las variables de tipo numérico, no hay mucho más que decir, serán aquellas que contienen valores numéricos, ya sean enteros o reales. Las variables lógicas presentan dos valores posibles, verdadero (TRUE) y falso (FALSE). Estos valores deben de escribirse con mayúsculas y, fuera de un contexto de programación, pueden abreviarse a T y F, respectivamente. Merece la pena comentar que lo que hace realmente este tipo es asignar una etiqueta a los valores 0 (es FALSE) y 1 (es TRUE). Este tipo de variables es el que aparece como resultado de las comparaciones lógicas:
x<-27 # asignamos un valor a x
x>3 # consultamos si x es mayor a 3, el resultado es TRUE[1] TRUECuando se hace una asignación, R asume que la variable será del tipo o clase de aquello que se le ha asignado. No importa si la variable ya se había usado de otra manera. Por ejemplo:
edad<-27 # asignación de un valor numérico a x
class(edad) # consulta del tipo que tiene x[1] "numeric"al asignar un valor numérico a edad, esta será una variable de tipo numérico, tal y como indica la función class(edad). Si ahora hacemos
edad<-"anciano"
class(edad)[1] "character"no se produce ningún error. Aunque antes fuera numérica, ahora edad ha cambiado su tipo para ser una variable de tipo character.
La distinción de clases desde el punto de vista informático guarda una relación poco refinada con los tipos estadísticos. Se puede asumir que las variables categóricas están representadas con la clase character y las numéricas con numeric y con eso es suficiente. Sin embargo, R es un lenguaje concebido para el análisis estadístico, así que se puede adaptar mejor a la concepción estadística. A continuación se revisa cómo manejar las clases de R según los tipos estadísticos definidos en la Sección 2.1.
Definir una variable que no sea numérica asignándole directamente la relación de valores, da como resultado una variable de tipo character, es decir, una variable cuyos niveles vienen expresados mediante un texto:
fuma <-c("si","si","no","si","no","no","si","si","no","no")
class(fuma)[1] "character"El tipo, o la clase, character alude al tipo informático de fuma (cómo debe ser tratada por el ordenador), pero esto no siempre es aprovechable como tipo estadístico (qué métodos de síntesis y de análisis se pueden usar con ella). Por ejemplo, si deseamos conocer las categorías o niveles de fuma, la función a utilizar es levels(), pero el resultado que devuelve esta función no es la lista esperada de categorías (“si”,“no”):
levels(fuma)NULLEl sistema, sencillamente, no reconoce la presencia de categorías. La definición de fuma como una variable categórica requiere declarar explícitamente que es de este tipo, lo que se hace mediante la función factor(). En el siguiente código se presenta, en primer lugar, esta asignación. En la segunda línea se consulta de qué tipo es la variable fuma, en la tercera, cuáles son sus niveles y finalmente el nivel de cada caso (que refleja al final la relación de niveles):
fuma <- factor(c("si","si","no","si","no","no","si","si","no","no"))
class(fuma)[1] "factor"levels(fuma)[1] "no" "si"fuma [1] si si no si no no si si no no
Levels: no siLos niveles de fuma han sido reconocidos, de forma automática por R, al crear esta variable. Dejarle este reconocimiento al sistema es rápido, pero no es lo más aconsejable. Por ejemplo, si hay algún error en la escritura del nivel asignado a un caso, esto originará que el valor erróneo sea considerado -incorrectamente- como un nivel adicional. Para evitar situaciones así (piense en un número elevado de casos), conviene especificar los niveles a través del parámetro levels de la función factor(). Veamos un ejemplo: en el siguiente código se especifica, mediante el parámetro levels, que los niveles de fuma son “no” y “si”, pero se ha cometido un error al escribir la categoría a la que pertenece el último caso (“nno”). Al solicitar, a continuación, cuáles son los niveles de fuma vemos que no aparece “nno” como tal
fuma <- factor(c("si","si","no","si","no","no","si","si","no","nno"),levels=c("no","si"))
levels(fuma)[1] "no" "si"Si vemos el listado de todos los casos, podemos comprobar que la categoría de fuma para el último es <NA> (valor faltante) poniendo de manifiesto que, en la definición original, no se asigno una categoría lícita a este paciente:
fuma [1] si si no si no no si si no <NA>
Levels: no siSiguiendo con nuestro conjunto de 10 casos, consideremos una nueva variable que refleje su estado de salud tras una última revisión clínica. Definimos entonces la variable estado, que va a ser de tipo cualitativo con las categorías “peor”, “igual” y “mejor”. Según lo visto en el apartado anterior, es inmediato definir estad usando a la función factor(). Conviene hacer también la declaración explícita de sus niveles con el argumento levels:
estado <- factor(c("igual","igual","peor","mejor","peor","igual","mejor","igual","mejor","mejor"),levels=c("peor","igual","mejor"))Hasta aquí todo bien, salvo el hecho de que el sistema no reconoce la relación ordinal existente entre los niveles. Es decir, la variable estado, tal y como ha sido definida es, desde el punto de vista estadístico, de tipo nominal. Podemos constatar este hecho consultando si existe la relación de orden a través de la función is.ordered():
is.ordered(estado)[1] FALSEVeamos dos formas equivalentes de establecer que una variable categórica sea de tipo ordinal. La primera es indicar ordered=TRUE como argumento de la función factor(). La segunda es definir la nueva variable utilizando la función ordered(), en lugar de factor(). Las dos alternativas generan el mismo resultado, una variable de tipo categórico con relación ordinal entre sus modalidades. El siguiente código ilustra las dos formas de proceder:
estado<-factor(c("igual","igual","peor","mejor","peor","igual","mejor","igual","mejor","mejor"),levels=c("peor","igual","mejor"),ordered=TRUE)
estado<-ordered(c("igual","igual","peor","mejor","peor","igual","mejor","igual","mejor","mejor"),levels=c("peor","igual","mejor"))Podemos ratificar el carácter ordinal de la variable estado consultando al sistema esta ordenada:
is.ordered(estado)[1] TRUEo, también, solicitando la clase a la que pertenece dicha variable
class(estado)[1] "ordered" "factor" Como vemos, estado es, en realidad, de dos clases (hereda las propiedades de cada una): factor y ordered, lo que supone que sigue siendo una variable de tipo factor, pero con relación ordinal entre sus categorías. Finalmente, comprobamos, al invocar directamente a la variable, que el sistema especifica los niveles indicando su relación:
estado [1] igual igual peor mejor peor igual mejor igual mejor mejor
Levels: peor < igual < mejorLas variables numéricas no necesitan de una declaración especial. Basta con asignar valores cuantitativos a una variable y su tipo será, por definición, numeric. Veamos un ejemplo:
edad<-c(19, 18.1, 22.9, 27.8)
class(edad)[1] "numeric"En la declaración edad, es irrelevante si los valores son enteros (discretos) o reales (continuos), y en general, se puede trabajar tranquilamente sin necesidad de hacer explícita esta distinción. En ocasiones, sí que puede ser interesante prescindir de los decimales. Para ello, podemos redondear los valores reales mediante la función round(). Esta función tiene dos argumentos, en el primero se indica la variable o el/los valor/es a redondear, y en el segundo el número de decimales deseado. Este argumento es opcional, si no se escribe nada, o se escribe cero, entonces se redondea el primer argumento al valor entero más próximo.
x<-c(1.132, 2.6598) # definición de un vector de ejemplo
round(x,2) # redondeo a dos decimales del vector x [1] 1.13 2.66round(x) # redondeo a un valor entero, sin decimales, del vector x[1] 1 3Otra forma de eliminar los decimales es truncar la variable. Esto se hace mediante la función trunc(), pero su uso debe estar bien justificado, por ejemplo para redondear siempre al alza y no al entero más cercano.
trunc(x) # truncado del vector x[1] 1 2trunc(x)+1 # redondeo al alza de x[1] 2 3Al redondear al entero más próximo, la clase de la variable no cambia, sigue siendo numeric. Sin embargo, existe la clase integer. Para forzar que una variable tenga este tipo se puede usar la función as.integer(). Es posible averiguar si una variable lo tiene mediante la función de consulta is.integer(). En el siguiente ejemplo comprobamos cómo eliminar los decimales de una variable real no la convierte en integer (sigue siendo numeric):
y<-trunc(x) # la variable y almacena el truncado del vector x
class(y) # y no es "integer", sigue siendo de tipo "numeric"[1] "numeric"z<-as.integer(y) # se define z forzando que sean valores enteros
z # el efecto es que se trunca y[1] 1 2class(z) # vemos que z es "integer"[1] "integer"is.integer(z) # la consulta confirma que lo es[1] TRUEis.numeric(z) # pero aunque sea "integer", z no deja de ser "numeric"[1] TRUETambién es posible convertir variables de texto en numéricas. Como esto puede parecer extraño, veámoslo con un ejemplo:
a<-c("1","2","3")
a[1] "1" "2" "3"class(a)[1] "character"Si probamos a ejecutar a*2, el programa mostrará un error indicando que el argumento no es numérico, la operación "1"+"1" es ilícita (no es lo mismo que 1+1 sin comillas). Como el contenido de la variable a guarda una relación unívoca con valores numéricos, sí que podemos hacer la conversión a auténticos números
x<-as.numeric(a)
x[1] 1 2 3class(x)[1] "numeric"Este tipo de operación puede ser necesaria en ocasiones en que se importan datos de otras aplicaciones y estos son leídos como texto. La función contraria para convertir un número en texto también existe: as.character().
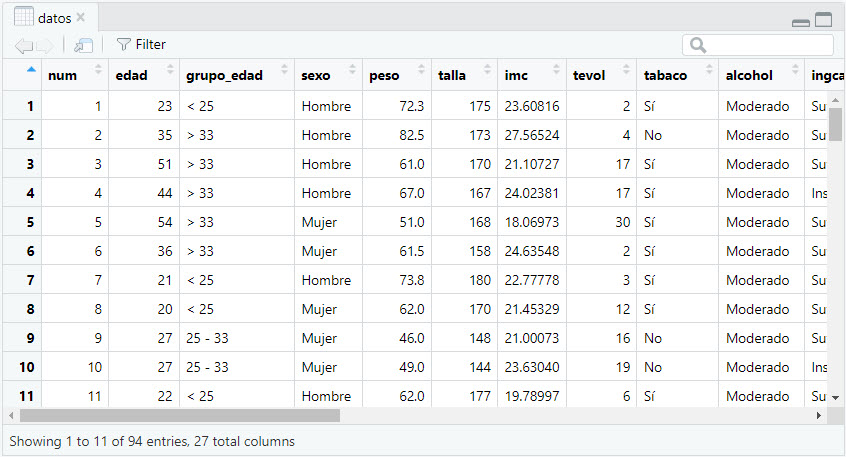
Por tabla o matriz de datos o base de datos entendemos la estructura de datos que R denomina data.frame (literalmente se traduciría como marco de datos). Un data.frame se presenta como un arreglo rectangular de los datos -similar a una matriz- en donde cada fila representa a un caso y cada columna se corresponde con una variable (en el ámbito estadístico, se alude a él como tabla de casos x variables). A diferencia de una matriz, en donde las filas y las columnas deben presentar valores de la misma clase (numéricos, alfanuméricos, …), en un data.frame, los elementos de una misma columna deben ser de la misma clase, pero los de columnas diferentes pueden pertenecer a clases también diferentes. Por ejemplo, en la Figura 3.5 se presenta un data.frame con cuatro filas (los casos) y seis columnas (las variables).
Una forma inmediata de crear un data.frame introduciendo toda la información desde el teclado es generar una base de datos vacía y editarla. En la edición se pueden definir, tanto los nombres y tipo de cada variable, como los valores que estas toman en cada caso. El código sería el siguiente:
datos<-data.frame() #creamos un data.frame vacío
edit(datos)Otra forma de crear un ´data.frame´ es a partir de la definición previa de las variables que lo van a conformar. Veamos un ejemplo, supongamos que deseamos crear la base de datos mostrada en la Figura 3.5 con la información de cuatro historias clínicas
historia<-I(c("C213/22","A4938/21","H342/23","M123/23")) #no hace falta la I()
edad<-c(25,50,44,37)
peso<-c(72.1,83.5,90.2,62.1)
talla<-c(1.85,1.70,1.69,1.75)
fuma<-factor(c("Si","No","No","No"))
estado<-ordered(c("peor","igual","mejor","mejor"),levels=c("peor","igual","mejor"))Para reunir todas estas variables en una base de datos, basta con escribir
pacientes<-data.frame(historia,edad,peso,talla,fuma,estado)Una cuestión inmediata, pero que conviene comentar dada su importancia, es que todas las variables deben de tener el mismo número de casos (en nuestro caso, cuatro), y que el primer valor de cada variable se corresponde al primer paciente, el segundo valor, al segundo paciente, y así hasta el final. Hay que reconocer que definir una base de datos de esta manera encierra cierto peligro cuando el número de casos es mayor. Lo más lógico sería introducir la información de todas las variables de cada paciente antes de pasar al siguiente, y no poner todos los valores del peso, todos los de la talla, etc.
Por estructura de una base de datos se alude a qué variables presenta y de qué tipo son. Esta información la podemos obtener a través de la función str():
str(pacientes)'data.frame': 4 obs. of 6 variables:
$ historia: 'AsIs' chr "C213/22" "A4938/21" "H342/23" "M123/23"
$ edad : num 25 50 44 37
$ peso : num 72.1 83.5 90.2 62.1
$ talla : num 1.85 1.7 1.69 1.75
$ fuma : Factor w/ 2 levels "No","Si": 2 1 1 1
$ estado : Ord.factor w/ 3 levels "peor"<"igual"<..: 1 2 3 3Como vemos, la función comienza indicando el hecho de que pacientes es un data.frame y sus dimensiones (4 observaciones de 6 variables). A continuación da la información pertinente de cada variable: historia es una variable con niveles arbitrarios, no identificados como niveles de un factor (esto es lo que significa AsIs) y de tipo carácter (chr). Las tres siguientes son numéricas (num). La variable fuma es un factor con 2 niveles (w/ 2) que son “No” y “Si”. Finalmente, estado es una variable ordinal (Ord.factor) con 3 niveles (w/ 3) que son “peor”<“igual”<“mejor”. Tras esta información, siempre aparecen los valores de los primeros casos (aquí solo son cuatro, si hubiera más observaciones, solo mostrará las primeras).
Las dimensiones de la base de datos se pueden obtener de forma individual mediante las siguientes funciones:
dim(pacientes) # devuelve el número de filas y de columnas[1] 4 6nrow(pacientes) # devuelve el número de filas[1] 4ncol(pacientes) # devuelve el número de columnas[1] 6length(pacientes) # es equivalente a ncol()[1] 6Los nombres de las variables que componen nuestra base de datos se pueden obtener a través de la función names() y también de colnames() (como vemos, es una tónica el hecho de que en R haya varias formas para conseguir un mismo fin)
colnames(pacientes)[1] "historia" "edad" "peso" "talla" "fuma" "estado" Puede ser interesante que nuestra base de datos también tenga nombres de fila, una etiqueta identificadora de cada caso. Cuando es así, la forma de consultar los nombres de fila es mediante la función rownames(). Pero esta función, al igual que ocurre con colnames(), sirve tanto para consultar como para establecer los valores correspondientes. Por ejemplo, supongamos que queremos nombrar cada fila de nuestra base de datos con las iniciales del paciente correspondiente. La forma de hacerlo es la siguiente:
rownames(pacientes)<-c("APC","PFF","MMA","JCS")Podemos comprobar el efecto de esta asignación utilizando la misma función rownames(), pero ahora a modo de consulta:
rownames(pacientes)[1] "APC" "PFF" "MMA" "JCS"Para ver la base de datos, no hay más que escribir su nombre, pero esto no es nada práctico cuando el número de casos es mayor -y mas interesante- que los cuatro casos que tenemos almacenados en pacientes. Las funciones head() y tail() permiten reproducir en pantalla la información de los primeros casos, la cabecera, y de los últimos respectivamente. En cada una de ellas, podemos escribir el número de casos que desamos ver. Por ejemplo, para hacer el listado con los dos primeros casos de pacientes escribimos
head(pacientes,2) historia edad peso talla fuma estado
APC C213/22 25 72.1 1.85 Si peor
PFF A4938/21 50 83.5 1.70 No igualy para hacerlo con los dos últimos casos
tail(pacientes,2) historia edad peso talla fuma estado
MMA H342/23 44 90.2 1.69 No mejor
JCS M123/23 37 62.1 1.75 No mejorSi omitimos el número de casos a presentar, el valor por defecto en ambas funciones es de seis.
La forma de aludir a cada una de las variables reunidas en un data.frame es utilizando el símbolo $ para separar el nombre del data.frame del nombre de la variable. Por ejemplo, si queremos ver los valores de la edad de nuestra base de datos pacientes no hay más que escribir
pacientes$edad[1] 25 50 44 37Una ventaja de usar RStudio, es que al escribir el nombre del data.frame seguido del símbolo $, se despliega un recuadro en el que podemos elegir el nombre de la variable que nos interesa seleccionándolo en la lista mostrada. Sin embargo, cuando todo el trabajo se concentra en una base de datos concreta, suele resultar un poco pesado ir escribiendo continuamente el nombre del data.frame seguido del $. Resultaría más práctico escribir el nombre de la variable y que R se diera por enterado de cuál es la base de datos que estamos manejando en la sesión de trabajo. Esto se puede hacer con el comando attach() (lo podemos traducir como vincular). Así, al escribir
attach(pacientes)The following objects are masked _by_ .GlobalEnv:
edad, estado, fuma, historia, peso, tallaya no es necesario escribir pacientes$ delante del nombre de cada variable. Cuando escribamos cualquier nombre -por ejemplo edad- R lo buscará en la base de datos vinculada -en este caso pacientes- y, si existe, asume que nos estamos refiriendo a esa variable -pacientes$edad-. Se pueden tener vinculadas diferentes bases de datos al mismo tiempo pero, cuidado, si un mismo nombre de variable aparece en más de un data.frame, uno de ellos enmascarará a los restantes. Una situación similar se da cuando en el entorno de trabajo hay definida una variable con el mismo nombre que el de una columna de un data.frame. Al vincular este último, su columna no será accesible; el sistema solo reconocerá a la variable previamente definida en el entorno. Como vemos, la vinculación de tablas no está exenta de posibles comportamientos indeseados, así que se debe controlar qué es lo que hay vinculado en todo momento. Cuando ya no sea necesaria la vinculación de una tabla, esta se puede desvincular utilizando la función detach():
detach(pacientes)Otra forma de aludir a las variables del data.frame sin tener que reiterar el prefijo <nombre del data.frame>$ es mediante el uso de la función with(). Su uso consiste en indicar el nombre del data.frame como primer argumento de la función y, como segundo argumento y entre corchetes, escribir todas las líneas de código que aludan a sus variables. Por ejemplo, el siguiente código permite definir la variable índice de masa corporal (calculado como el peso -en kg- dividido por la talla -en metros- elevada al cuadrado) a partir de los datos de pacientes utilizando with():
with(pacientes, {
IMC=peso/talla^2 # calcula el índice de masa corporal
IMC # muestra sus valores
})[1] 21.06647 28.89273 31.58153 20.27755El código ha realizado su trabajo y lo hemos hecho sin tener que escribir pacientes$peso y pacientes$talla, pero la nueva variable IMC ya no permanece accesible. Para poder mantenerla debemos asignarla a una variable del entorno externo al ámbito de with(). Es más, podemos incorporar a IMC en el data.frame pacientes tal y como se presenta a continuación:
pacientes$IMC<- with(pacientes, {
IMC=peso/talla^2 # calcula el índice de masa corporal
IMC # muestra sus valores
})Este código permite ilustrar una forma de añadir nuevas variables a un data.frame, basta con asignarles valores como si ya existieran. El sistema las creará y asignará los valores indicados.
Si queremos obtener la información correspondiente al segundo paciente en la tercera variable del data.frame, basta con escribir el nombre del data.frame y la posición deseada. La sentencia
pacientes[2,3][1] 83.5devuelve el valor del peso del segundo paciente. Es decir, con la sintáxis
obtenemos la información correspondiente a las filas y columnas indicados (los signos <> delimitan qué es lo que se debe escribir en su lugar, no hay que escribirlos). La generalización del ejemplo anterior a más de una fila y a más de una variable es, por ejemplo:
pacientes[2:3,3:5] peso talla fuma
PFF 83.5 1.70 No
MMA 90.2 1.69 NoCon esta sentencia se ha pedido la información de los pacientes que ocupan las filas 2 y 3, relativa a las variables que ocupan las columnas 3, 4 y 5 (elpeso, la talla y si fuma). También se puede solicitar la información de casos o de variables que no estén seguidos indicando las posiciones mediante un vector (usando c()). Por ejemplo, el siguiente código muestra la edad (2ª variable) y el estado (6ª variable) de los casos que ocupan las filas 1 y 3:
pacientes[c(1,3),c(2,6)] edad estado
APC 25 peor
MMA 44 mejorNo es muy cómodo tener que mirar en qué número de columna está cada variable, así que una sintáxis alternativa, y más intuitiva es:
pacientes[c(1,3),c("edad","peso")] edad peso
APC 25 72.1
MMA 44 90.2Finalmente, si lo que deseamos es obtener la información relativa a todas las variables para unos casos concretos, basta dejar el hueco -dentro del corchete- reservado a las columnas vacío. Por ejemplo, toda la información relativa al segundo caso se obtiene como sigue:
pacientes[2, ] historia edad peso talla fuma estado IMC
PFF A4938/21 50 83.5 1.7 No igual 28.89273Con esta regla, podemos pensar que si escribimos pacientes[,], el resultado debe de ser el listado de todos los casos (la posición de las filas está vacía) con todas las variables (la posición de las columnas está vacía). Efectivamente, es así, solo que no hace falta escribir los corchetes y la coma, basta con poner pacientes a secas y el resultado es el mismo.
Quizá, mas habitual que seleccionar los casos por el número de fila que ocupan, sea hacerlo por que cumplan o no una condición. Por ejemplo, que la edad sea mayor a 40 años o que sean fumadores (fuma="Si"). Para seleccionar todas las variables de aquellos casos que cumplen una condición, basta utilizar la sintaxis precedente, solo que en lugar del número de fila, escribimos la condición. Las condiciones son sentencias lógicas, es decir, relaciones entre variables que devuelven el valor verdadero (TRUE) si se da la relación, o falso (FALSE) en caso contrario. En la elaboración de sentencias lógicas intervienen dos tipos de operadores: los operadores relacionales y los operadores lógicos. Veamos algo de ellos.
Operadores relacionales Mediante este tipo de operadores se elaboran las sentencias lógicas simples. Los operadores relacionales establecen -como su nombre indica- el tipo de relación entre una variable y un valor, o entre dos variables. Estos operadores son
| Operador | Relación | Operador | Relación |
|---|---|---|---|
| == | Igual | != | diferente |
| < | menor | > | mayor |
| <= | menor o igual | >= | mayor o igual |
La sintaxis con este tipo de operadores debe de obedecer a una regla similar a esta (OR alude al operador relacional)
Por ejemplo, edad > 40 es una sentencia lógica simple. Si en nuestra base de datos tuvieramos dos medidas del peso, por ejemplo, antes (peso1) y después (peso2) de un periodo de tratamiento, podríamos elaborar la sentencia lógica simple peso1 > peso2 para detectar a aquellos pacientes que han perdido peso, o también peso1 >= peso2 para aquellos que no han variado o lo han reducido.
Veamos el uso de sentencias lógicas simples para seleccionar, o filtrar, casos. Para seleccionar a todos los casos cuyo peso es superior a 80 kg, escribimos
pacientes[peso>80,] historia edad peso talla fuma estado IMC
PFF A4938/21 50 83.5 1.70 No igual 28.89273
MMA H342/23 44 90.2 1.69 No mejor 31.58153Podemos asignar este resultado a una nueva variable y así tendríamos un nuevo data.frame con la selección realizada:
sobrepeso<-pacientes[peso>80,]El resultado es que hemos definido un nuevo data.frame cuyo contenido es el subconjunto de pacientes que pesan más de 80 kg. Y hemos incluido en él a todas las variables, ya que hemos dejado en blanco el espacio reservado para indicar las columnas después de la coma.
Cuando una sentencia lógica se va a utilizar más, conviene guardarla como variable. El siguiente código da el mismo resultado que el anterior
condicion<-peso>80
sobrepeso<-pacientes[condicion,]
sobrepeso historia edad peso talla fuma estado IMC
PFF A4938/21 50 83.5 1.70 No igual 28.89273
MMA H342/23 44 90.2 1.69 No mejor 31.58153Pero ahora, tenemos la sentencia lógica almacenada en la variable condicion, de manera que la podemos utilizar más veces. Por cierto, obsérvese que el código anterior ha creado un nuevo data.frame con nombre sobrepeso y que contiene a todos los casos de pacientes que cumplen con el criterio dado en condicion. Pero ¿de qué clase es la variable condicion? Lo averiguamos fácilmente:
class(condicion)[1] "logical"| Operador lógico | símbolo | Tabla lógica |
|---|---|---|
| Y | & | TRUE & TRUE = TRUE TRUE & FALSE = FALSE FALSE & FALSE = FALSE |
| O | | | TRUE | TRUE = TRUE TRUE | FALSE = TRUE FALSE | FALSE = FALSE |
| No (negación) | ! | ! FALSE = TRUE ! TRUE = FALSE |
Las sentencias lógicas simples se pueden combinar para dar lugar a sentencias lógicas más complejas. Aquí es donde intervienen los operadores lógicos, que básicamente son los siguientes:
La sintaxis es la siguiente (OL alude al operador lógico)
Por ejemplo, para seleccionar a los pacientes que pesan más de 80 kg y fuman escribiríamos
condicion <- (peso>80) & (fuma=="Si")
pacientes[condicion,][1] historia edad peso talla fuma estado IMC
<0 rows> (or 0-length row.names)Es buena práctica encerrar a las sentencias lógicas simples entre paréntesis. Si ahora queremos seleccionar a los pacientes que fuman o que pesan más de 80 kg, escribiremos
pacientes[(peso>80)|(fuma=="Si"),] historia edad peso talla fuma estado IMC
APC C213/22 25 72.1 1.85 Si peor 21.06647
PFF A4938/21 50 83.5 1.70 No igual 28.89273
MMA H342/23 44 90.2 1.69 No mejor 31.58153Es importante mantener la sintaxis descrita. Veamos un último ejemplo. Si queremos seleccionar aquellos casos con una edad comprendida entre 20 y 40 años, no podemos escribir como condición 20<edad<40, esto no funciona. Se deben elaborar las dos sentencias simples edad>20 y edad<40 y relacionarlas con el operador lógico y tal y como se presenta a continuación:
pacientes[(edad>20)&(edad<40),] historia edad peso talla fuma estado IMC
APC C213/22 25 72.1 1.85 Si peor 21.06647
JCS M123/23 37 62.1 1.75 No mejor 20.27755Se pueden añadir nuevos casos (filas) a un data.frame utilizando la función rbind() (de row bind, unir filas). Para ello, se debe crear el registro del nuevo caso asignando los valores correspondientes a las variables definidas en el data.frame. Cuando no se conoce el valor de alguna variable presente en el data.frame, se debe asignar el código de valor faltante NA A continuación, se debe indicar, como argumentos de rbind(), el nombre del data.frame y el de la fila a añadir. El resultado de rbind() se debe asignar al data.frame original, o bien a otro, por que si no se asinga, la fusión se hace pero no se conserva. Por ejemplo, en el código siguiente se añade un nuevo paciente a pacientes, el resultado se muestra en pantalla, pero el data.frame pacientes no lo conserva:
# definición del nuevo paciente
nuevo_pac<-c(historia="HML/23",
edad=39,
peso=82.5,
talla=1.80,
fuma="Si",
estado="peor",
IMC=NA
)
rbind(pacientes,nuevo_pac) historia edad peso talla fuma estado IMC
APC C213/22 25 72.1 1.85 Si peor 21.0664718772827
PFF A4938/21 50 83.5 1.7 No igual 28.8927335640138
MMA H342/23 44 90.2 1.69 No mejor 31.581527257449
JCS M123/23 37 62.1 1.75 No mejor 20.2775510204082
5 HML/23 39 82.5 1.8 Si peor <NA>pacientes historia edad peso talla fuma estado IMC
APC C213/22 25 72.1 1.85 Si peor 21.06647
PFF A4938/21 50 83.5 1.70 No igual 28.89273
MMA H342/23 44 90.2 1.69 No mejor 31.58153
JCS M123/23 37 62.1 1.75 No mejor 20.27755La forma de incorporar realmente al nuevo caso al data.frame deseado es
pacientes<-rbind(pacientes,nuevo_pac)Para ordenar los registros de un data.frame, basta con referenciar sus filas mediante los elementos de un vector con el orden deseado. Por ejemplo, el código siguiente muestra los registros de pacientes en el orden establecido en el vector listado:
pacientes # se muestra el orden original de las filas historia edad peso talla fuma estado IMC
APC C213/22 25 72.1 1.85 Si peor 21.06647
PFF A4938/21 50 83.5 1.70 No igual 28.89273
MMA H342/23 44 90.2 1.69 No mejor 31.58153
JCS M123/23 37 62.1 1.75 No mejor 20.27755listado<-c(3,2,4,1) # vector de indices
pacientes[listado,] # ahora el orden de las filas es el establecido por el vector historia edad peso talla fuma estado IMC
MMA H342/23 44 90.2 1.69 No mejor 31.58153
PFF A4938/21 50 83.5 1.70 No igual 28.89273
JCS M123/23 37 62.1 1.75 No mejor 20.27755
APC C213/22 25 72.1 1.85 Si peor 21.06647Si deseamos conservar ese orden, bastaría con asignar la salida a ese mismo data.frame, o a uno nuevo. Como las filas están etiquetadas, también podemos hacer
listado<-c('PFF','MMA','APC','JCS')
pacientes[listado,] historia edad peso talla fuma estado IMC
PFF A4938/21 50 83.5 1.70 No igual 28.89273
MMA H342/23 44 90.2 1.69 No mejor 31.58153
APC C213/22 25 72.1 1.85 Si peor 21.06647
JCS M123/23 37 62.1 1.75 No mejor 20.27755Aunque el interés de esto puede ser más relativo. En general, ordenar los registros de un data.frame tiene por cometido el de realizar consultas. Por ejemplo, con frecuencia interesa saber qué caso es el que tiene el valor mínimo, o el máximo, en una variable determinada. Para identificar ese caso, lo más rápido suele ser ordenar las filas del data.frame, en sentido creciente o decreciente respectivamente, de dicha variable. Supongamos que deseamos saber quién es, qué peso tiene y si fuma el paciente con mayor peso del data.frame pacientes. Ordenando dicha base de datos en sentido decreciente de la variable peso, el primer registro deberá presentar el caso con mayor peso (o los casos, si es que ese valor máximo se repite). La función order() permite hacer esto. Como primer argumento de order() se debe indicar la variable que sirve como criterio de ordenación. El argumento lógico decreasing permite especificar si el orden es de menor a mayor o al revés (su valor por defecto es FALSE, por lo que si no se especifica nada, ordena de menor a mayor). Sin embargo, el resultado de order() no es exactamente el data.frame ordenado. Lo que proporciona esta función es el orden en que se deben organizar las filas de la base de datos para cumplir con el criterio especificado (es el vector lista usado en el ejemplo del párrafo anterior). Veamos un ejemplo en el que se trata de ordenar a los casos de pacientes según valores decrecientes de la variable peso:
order(pacientes$peso, decreasing=T)[1] 3 2 1 4Este resultado debe interpretarse como que el caso con mayor peso ocupa la fila 3 del data.frame, el siguiente la fila 2, etc. Ahora podríamos escribir pacientes[3,] para obtener toda la información acerca del paciente con mayor peso. Pero si lo que se pretende es ordenar al conjunto de datos, la información devuelta por order() se debe utilizar como índice de filas de la siguiente manera:
# obtenemos el vector con la ordenación y lo asignamos a una variable 'orden'
orden<-order(pacientes$peso, decreasing=T)
# utilizamos 'orden' para establecer el orden de las filas de 'pacientes'
pacientes[orden,] historia edad peso talla fuma estado IMC
MMA H342/23 44 90.2 1.69 No mejor 31.58153
PFF A4938/21 50 83.5 1.70 No igual 28.89273
APC C213/22 25 72.1 1.85 Si peor 21.06647
JCS M123/23 37 62.1 1.75 No mejor 20.27755El código anterior solo muestra el contenido de pacientes ordenado por valores decrecientes del peso, pero no altera el orden en el propio data.frame. Si se desea conservar este orden establecido, hay que asignar el resultado, bien al propio data.frame, o bien creando uno nuevo:
pacientes<-pacientes[orden,]La función order() admite especificar más de una clave de ordenación. La primera que se indica es la que establece el orden, pero cuando dos casos presentan el mismo valor para esta variable, se utiliza el criterio indicado en segundo lugar. El planteamiento admite la consideración de cualquier número de variables como criterio. El sentido de la ordenación debe especificarse para cada una asignando el correspondiente vector de valores lógicos al argumento decreasing. En el siguiente ejemplo, se ordena según el orden decreciente de la variable estado. Cuando hay más de un caso con el mismo estado, el orden lo impone la edad en sentido creciente:
orden<-order(pacientes$estado,pacientes$edad,decreasing = c(T,F))
pacientes[orden,] historia edad peso talla fuma estado IMC
JCS M123/23 37 62.1 1.75 No mejor 20.27755
MMA H342/23 44 90.2 1.69 No mejor 31.58153
PFF A4938/21 50 83.5 1.70 No igual 28.89273
APC C213/22 25 72.1 1.85 Si peor 21.06647Obviamente, se podría haber utilizado directamente la función order() directamente como primer argumento dentro del corchete de la segunda línea, sin recurrir a la creación de la variable intermediaria orden. Pero esto complica la sentencia innecesariamente. Además, la creación de la variable orden permite volver a utilizar este criterio de ordenación más adelante si es necesario.
Ya vimos cómo crear una nueva variable IMC e incluirla en la base de datos pacientes. Basta con definir la variable y asignarla al mismo como nueva columna:
pacientes$IMC<-pacientes$peso/pacientes$talla^2Por recodificación se entiende el cambio en los valores actuales de una variable por otros que se han de especificar. En el caso de
Esta reasignación de valores se puede almacenar en la propia variable de origen o bien en una variable nueva. Veamos varias posibilidades.
Recodificación de factores. Cuando se considera una variable de tipo factor, y lo que se desea es cambiar sus etiquetas, la recodificación es inmediata. Basta con utilizar la función factor() (o, en su caso, ordered()) con una nueva especificación en el argumento labels. A continuación se ilustra esta idea con un ejemplo en el que se trata de cambiar los valores de la variable estado (peor, igual, mejor) a solo sus iniciales (p, i, m). Para ello creamos una nueva variable, por ejemplo estado2, que recibirá los nuevos valores. Seguidamente, se pueden asignar los valores de estado2 al data.frame, como una nueva columna o reemplazando los valores de estado:
# hacemos el cambio de etiquetas asignándolo a una nueva variable
est<-ordered(pacientes$estado, labels=c("p","i","m"))
# si se desea añadir estado2 como una nueva columna, escribimos
pacientes[,"estado2"]<-estado2
# o también
pacientes<-data.frame(pacientes,estado2)
# si lo se desea es reemplazar la variable estado del data.frame por
# la nueva codificación, basta con hacer
pacientes$estado<-estado2Transformación de variables cuantitativas. El hecho de considerar una variable auxiliar (como estado2 en el ejemplo anterior) al recodificar, es muy recomendable para no perder la información original. Así, si por ejemplo se comete un error en la especificación y el resultado se almacena en otra variable, es fácil reparar el error ya que contamos con la información de partida. En el ejemplo siguiente, se obtiene los valores de la talla expresados en centímetros:
pacientes2<-pacientes
pacientes2$estado[pacientes$estado=="peor"]<-"empeora"Warning in `[<-.factor`(`*tmp*`, pacientes$estado == "peor", value =
structure(c(NA, : invalid factor level, NA generatedLa función cut() permite especificar puntos de corte para definir categorías a partir de los valores de una variable numérica.
El argumento breaks permite establecer los puntos de corte bajo dos criterios diferentes. El primero, es indicar en cuántos grupos se desea agrupar la variable cuantitativa. Para ello, basta asignar un único valor numérico a breaks. Por ejemplo, el siguiente código permite dicotomizar a la variable edad:
pacientes[,"gedad"] <- cut(pacientes$edad, breaks = 2)
# presentamos solo las tres columnas indicadas:
pacientes[ , c("historia","edad","gedad")] historia edad gedad
APC C213/22 25 (25,37.5]
PFF A4938/21 50 (37.5,50]
MMA H342/23 44 (37.5,50]
JCS M123/23 37 (25,37.5]Observemos que el punto de corte, que se establece de forma automática, es aquel que divide a la amplitud de la variable en dos partes iguales, es decir, la media aritmética entre los dos extremos observados \[corte=\frac{min(<variable>)+max(<variable>)}{2}\] En el caso de la edad este valor es de 37.5. El mismo razonamiento es válido cuando se trata de más grupos; el recorrido de la variable se divide en aquel número de intervalos de la misma amplitud que indique el argumento breaks. También podemos observar en el ejemplo anterior, cómo a cada grupo se asigna una etiqueta (label) informativa del intervalo elaborado. El criterio en la elaboración de dichos intervalos es que el límite inferior no entra en el intervalo (esto se indica con un paréntesis, se dice que el intervalo es abierto por la izquierda) y el superior sí (es lo que indica el corchete, se dice que el intervalo es cerrado por la derecha). Según este criterio, los casos cuyo valor de la variable a categorizar coincida con el mínimo, serán excluidos y no pertenecerán a ningún grupo. Esto se evita automáticamente considerando que el primero de los intervalos es cerrado por la izquierda (su etiqueta debería indicar [25,37.5]). Esta regla de que los intervalos sean cerrados por la derecha o por la izquierda se puede modificar con el argumento right de cut(), que por defecto tiene el valor TRUE.
El segundo criterio de categorización que se puede asumir es indicando, de forma explícita, los límites deseados para los intervalos que definen cada categoría. Esto se hace también a través del parámetro breaks, que ahora adoptará forma vectorial. Veamos un ejemplo:
pacientes[,"gedad"] <- cut(pacientes$edad, breaks = c(20,30,40,50))
pacientes[,c("historia","edad","gedad")] historia edad gedad
APC C213/22 25 (20,30]
PFF A4938/21 50 (40,50]
MMA H342/23 44 (40,50]
JCS M123/23 37 (30,40]Al indicar cuatro valores en breaks, se está diciendo que el primero sea el límite inferior del primer intervalo y el último el límite superior del último. Los valores intermedios indican los puntos de corte que constituyen la frontera entre los intervalos a elaborar entre estos dos extremos. Conviene mencionar que ahora cut() no contempla que el primer intervalo sea cerrado por la izquierda. Si fuera así, el mínimo observado nunca sería incluido en alguna categoría. Vamos a comprobarlo, para ello establecemos un criterio diferente en breaks:
pacientes[,"gedad"] <- cut(pacientes$edad, breaks = c(25,35,45,55))
pacientes[,c("historia","edad","gedad")] historia edad gedad
APC C213/22 25 <NA>
PFF A4938/21 50 (45,55]
MMA H342/23 44 (35,45]
JCS M123/23 37 (35,45]Para solucionar esto, cut() contempla el argumento include.lowest, que por defecto tiene el valor FALSE. Basta con asignar include.lowest=T para que el primer intervalo sea cerrado por la izquierda (y por la derecha). Otra cuestión es que, a menudo, los intervalos inicial y final constituyen un cajón de sastre en donde incluir los extremos, más o menos dispersos, de la distribución de la variable a categorizar (suelen resultar no homogéneos en amplitud respecto a los demás intervalos). Es inmediato automatizar esto sustituyendo los valores primero y último del vector breaks por las funciones min() y max(), tal y como se ilustra a continuación (observe que, al especificar include.lowest=T, el primer intervalo ahora sí que se presenta cerrado por la izquierda):
pacientes[,"gedad"] <- cut(pacientes$edad, breaks = c(min(edad),35,45,max(edad)), include.lowest=T)
pacientes[,c("historia","edad","gedad")] historia edad gedad
APC C213/22 25 [25,35]
PFF A4938/21 50 (45,50]
MMA H342/23 44 (35,45]
JCS M123/23 37 (35,45]Como hemos visto, cut() asigna como etiqueta a cada intervalo la especificación de sus límites. Sin embargo, esto se puede modificar mediante el argumento labels, que deberá ser un vector de valores de texto con la misma longitud del vector dado en breaks.
pacientes[,"gedad"] <- cut(pacientes$edad,
breaks = c(min(edad),35,45,max(edad)),
labels = c("grupo 1", "grupo 2", "grupo 3"),
include.lowest=T)
pacientes[,c("historia","edad","gedad")] historia edad gedad
APC C213/22 25 grupo 1
PFF A4938/21 50 grupo 3
MMA H342/23 44 grupo 2
JCS M123/23 37 grupo 2Un último detalle. La variable obtenida a través de cut() es de tipo factor:
class(pacientes$gedad)[1] "factor"Sin embargo, parece lógico que si se está categorizando a una variable cuantitativa, el resultado debería ser una variable ordinal (ordered). Estableciendo ordered_result=T como argumento de cut(), se obtiene este resultado:
pacientes[,"gedad"] <- cut(pacientes$edad,
breaks = c(min(edad),35,45,max(edad)),
labels=c("grupo 1", "grupo 2", "grupo 3"),
ordered_result = T,
include.lowest=T)
class(pacientes$gedad)[1] "ordered" "factor"